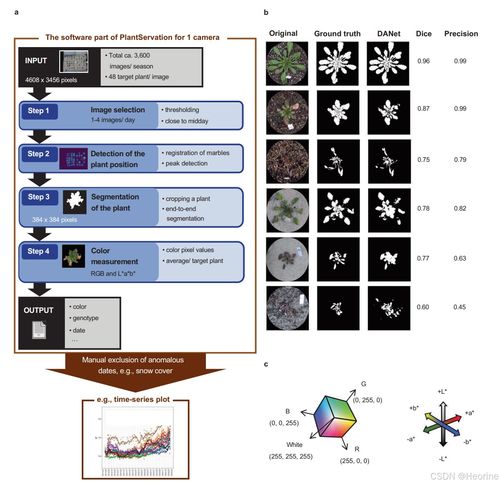
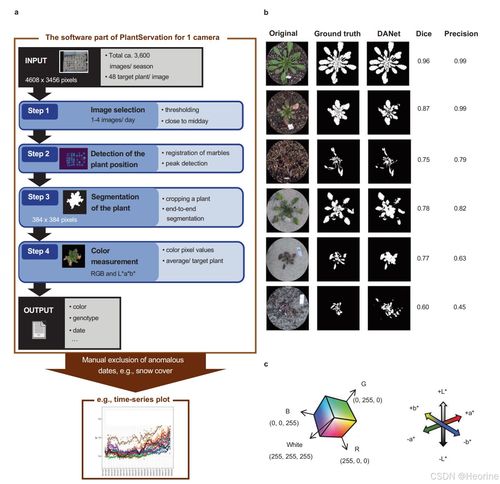
大家好,我是小李,最近在研究生物信息学的过程中,偶然发现了一篇Nature Plants上的论文,里面用到了一种非常酷炫的图表——变种火山图。作为一个数据可视化爱好者,我立刻被这种图表吸引住了。今天,我就来和大家分享一下如何使用R语言中的ggplot2包来绘制这种图表。
首先,我们先了解一下什么是变种火山图。变种火山图(Variant Volcano Plot)是一种结合了火山图和基因变异信息的图表,通常用于展示基因表达差异与变异频率之间的关系。它不仅可以直观地展示基因表达的变化,还可以通过颜色和点的大小来表示不同类型的变异,使得数据更加丰富和直观。
接下来,我们来看看如何用R语言中的ggplot2包来绘制这种图表。如果你还没有安装ggplot2包,可以通过以下命令进行安装:
install.packages("ggplot2")安装完成后,加载ggplot2包:
library(ggplot2)为了更好地理解如何绘制变种火山图,我们需要准备一些数据。假设我们有一个包含基因表达差异和变异频率的数据集,数据格式如下:
| Gene | logFC | pvalue | variant_type |
|---|---|---|---|
| Gene1 | 2.5 | 0.001 | Synonymous |
| Gene2 | -1.8 | 0.05 | Nonsynonymous |
| Gene3 | 1.2 | 0.0001 | Stopgain |
在这个数据集中,Gene是基因名称,logFC是基因表达的对数倍数变化,pvalue是显著性p值,variant_type是变异类型。我们可以根据这些数据来绘制变种火山图。
首先,我们导入数据并进行一些预处理。假设数据存储在一个CSV文件中,可以使用以下代码读取数据:
data <- read.csv("data.csv")接下来,我们需要对数据进行一些处理,例如计算-log10(pvalue),以便在图表中更好地展示p值。同时,我们还可以根据p值设置一个显著性阈值,例如0.05,并将显著的点标记出来:
data$log_pvalue <- -log10(data$pvalue) data$significant <- ifelse(data$pvalue < 0.05, "Significant", "Not Significant")现在,我们已经准备好绘制变种火山图了。我们可以使用ggplot2的geom_point()函数来绘制散点图,并通过aes()函数映射不同的变量到图表的不同属性上。例如,我们可以将logFC映射到x轴,log_pvalue映射到y轴,variant_type映射到颜色,significant映射到形状:
ggplot(data, aes(x = logFC, y = log_pvalue, color = variant_type, shape = significant)) + geom_point(size = 2) + theme_minimal() + labs(title = "Variant Volcano Plot", x = "Log Fold Change", y = "-log10(P-value)") + scale_color_manual(values = c("Synonymous" = "blue", "Nonsynonymous" = "red", "Stopgain" = "green")) + scale_shape_manual(values = c("Significant" = 16, "Not Significant" = 4))这段代码会生成一个漂亮的变种火山图,其中不同类型的变异用不同的颜色表示,显著的点用实心圆表示,非显著的点用空心圆表示。你可以根据自己的需求调整颜色、形状和大小,使图表更加个性化。
除了基本的散点图,我们还可以在图表中添加一些额外的元素,例如参考线和注释。例如,我们可以添加一条水平线来表示p值的显著性阈值:
ggplot(data, aes(x = logFC, y = log_pvalue, color = variant_type, shape = significant)) + geom_point(size = 2) + theme_minimal() + labs(title = "Variant Volcano Plot", x = "Log Fold Change", y = "-log10(P-value)") + scale_color_manual(values = c("Synonymous" = "blue", "Nonsynonymous" = "red", "Stopgain" = "green")) + scale_shape_manual(values = c("Significant" = 16, "Not Significant" = 4)) + geom_hline(yintercept = -log10(0.05), linetype = "dashed", color = "gray")这条虚线可以帮助读者更直观地理解哪些点是显著的。此外,我们还可以在图表中添加注释,突出显示某些重要的基因或区域:
ggplot(data, aes(x = logFC, y = log_pvalue, color = variant_type, shape = significant)) + geom_point(size = 2) + theme_minimal() + labs(title = "Variant Volcano Plot", x = "Log Fold Change", y = "-log10(P-value)") + scale_color_manual(values = c("Synonymous" = "blue", "Nonsynonymous" = "red", "Stopgain" = "green")) + scale_shape_manual(values = c("Significant" = 16, "Not Significant" = 4)) + geom_hline(yintercept = -log10(0.05), linetype = "dashed", color = "gray") + geom_text(aes(label = ifelse(abs(logFC) > 2 & log_pvalue > 3, Gene, "")), vjust = 1.5, hjust = 0.5)这段代码会在图表中标注出那些logFC绝对值大于2且log_pvalue大于3的基因,帮助读者快速找到重要的基因。
最后,为了让图表更加美观,我们还可以对主题进行一些调整。例如,使用theme()函数来自定义背景颜色、字体大小等:
ggplot(data, aes(x = logFC, y = log_pvalue, color = variant_type, shape = significant)) + geom_point(size = 2) + theme_minimal() + labs(title = "Variant Volcano Plot", x = "Log Fold Change", y = "-log10(P-value)") + scale_color_manual(values = c("Synonymous" = "blue", "Nonsynonymous" = "red", "Stopgain" = "green")) + scale_shape_manual(values = c("Significant" = 16, "Not Significant" = 4)) + geom_hline(yintercept = -log10(0.05), linetype = "dashed", color = "gray") + geom_text(aes(label = ifelse(abs(logFC) > 2 & log_pvalue > 3, Gene, "")), vjust = 1.5, hjust = 0.5) + theme(panel.background = element_rect(fill = "lightgray"), plot.title = element_text(size = 16, face = "bold"), axis.text = element_text(size = 12), axis.title = element_text(size = 14))通过这些调整,我们可以让图表更加美观和专业。当然,这只是绘制变种火山图的一个基础示例,实际应用中可以根据具体的需求进行更多的定制和优化。
总的来说,变种火山图是一种非常强大的工具,能够帮助我们在基因表达和变异分析中获得更多的洞见。通过使用R语言的ggplot2包,我们可以轻松地绘制出高质量的变种火山图,并根据自己的需求进行个性化的调整。希望这篇文章能够帮助你掌握这一技能,让你在数据分析中更加得心应手!

发表评论 取消回复