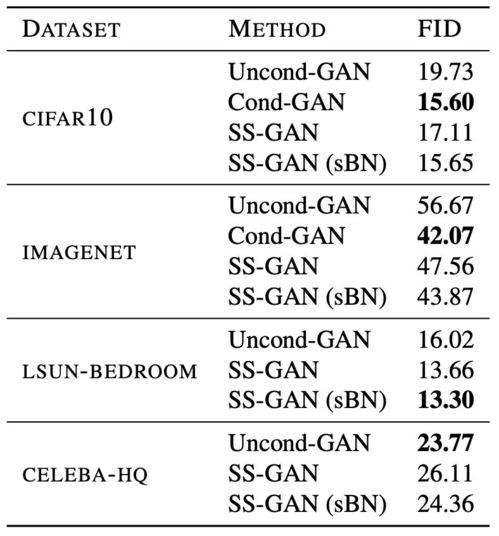
在基因组学研究的浩瀚星空中,小李是一位专注于生物信息学领域的探索者。他最近接触到了一个令人兴奋的新工具——GeneToCN,这是一种直接从NGS(下一代测序)数据中估计基因拷贝数的alignment-free方法。今天,让我们跟随小李的脚步,一起深入了解这项技术。
什么是GeneToCN?
小李首先解释了GeneToCN的核心概念。与传统依赖序列比对的方法不同,GeneToCN通过分析原始读取数据中的k-mer频率分布来推断基因拷贝数。这种方法的优势在于无需进行耗时的序列比对操作,从而显著提高了计算效率和适用性。
为什么选择GeneToCN?
小李指出,传统的基因拷贝数估计方法通常需要将测序数据映射到参考基因组上,这一过程不仅耗时,还可能受到参考基因组质量的影响。而GeneToCN采用无比对的方式,避免了这些问题,使得它在处理低质量或非模型物种的数据时表现出色。
实际应用案例
为了更好地理解GeneToCN的实际应用,小李分享了一个有趣的案例。在一项关于肿瘤异质性的研究中,研究人员使用GeneToCN成功地从大量样本中快速估计了关键驱动基因的拷贝数变化。这种高效且准确的分析为后续的功能验证提供了重要线索。
技术细节解析
小李深入探讨了GeneToCN的技术细节。该方法基于k-mer计数构建基因特异性特征向量,并利用机器学习模型对其进行分类和回归分析。最终输出的结果是每个目标基因的拷贝数估计值。这种创新的设计让GeneToCN成为基因组学研究中的强大工具。
未来展望
站在科技前沿的小李对GeneToCN的未来发展充满期待。他认为,随着测序技术的进步和算法优化,GeneToCN有望进一步提升其精度和速度,为个性化医疗、癌症研究等领域带来更多突破。
总结
GeneToCN作为一项革命性的技术,正在改变我们对基因拷贝数的理解方式。通过小李的分享,我们不仅了解了它的原理和优势,还看到了它在实际研究中的巨大潜力。如果你也对基因组学感兴趣,不妨尝试一下这个强大的工具吧!
文章导读:


发表评论 取消回复