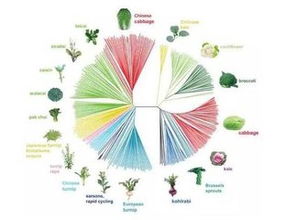
在这个科技飞速发展的时代,系统发育树的研究早已不再局限于枯燥的数据分析。作为一名热爱探索生命奥秘的人,我最近在简书上发现了一个热搜话题——“系统发育树的构建与优化”。这让我对这个领域的最新研究产生了浓厚的兴趣。
正如科技日报所报道的那样,西班牙基因组调控研究中心的科学家们在《自然·通讯》上发表了一项创新研究。他们提出了一种全新的方法,通过利用蛋白质的三维形状来解析生命的演化历程。这项技术不仅改变了我们对生物进化的理解,还为系统发育树的构建提供了全新的视角。
从序列选择开始
构建一棵系统发育树的第一步是序列的选择。这一步看似简单,实则充满了挑战。我们需要从海量的基因数据中筛选出具有代表性的序列,确保它们能够准确反映物种之间的亲缘关系。在我的实践中,我发现这一过程需要结合多种工具和算法,比如BLAST和Clustal Omega等。这些工具不仅可以帮助我们快速找到相似的序列,还能提供初步的比对结果。
进化模型的选择
有了合适的序列后,接下来就是选择合适的进化模型。不同的模型适用于不同的数据类型和研究目的。例如,Jukes-Cantor模型适合用于简单的核苷酸替换分析,而GTR模型则更适合复杂的多序列比对。在这个过程中,我会根据数据的特点和研究目标,灵活选择最适合的模型。有时候,甚至需要尝试多种模型,才能找到最理想的解决方案。
树的构建与优化
当序列和模型都准备就绪后,就可以开始构建系统发育树了。常用的软件包括MEGA、RAxML和MrBayes等。每种软件都有其独特的优点和局限性。在我的经验中,MEGA适合初学者使用,因为它界面友好且功能强大;而RAxML和MrBayes则更适合高级用户,因为它们提供了更多的参数选项和更高的计算效率。
构建完成后,还需要对树进行优化。这一步骤通常涉及树拓扑结构的调整和分支长度的校正。为了确保结果的可靠性,我常常会进行多次重复实验,并结合Bootstrap分析来评估节点的支持度。
进化树的美化
最后一步是进化树的美化。虽然科学数据本身已经足够吸引人,但一份精心设计的可视化图表可以让研究成果更加直观易懂。在这里,我可以推荐几款非常实用的工具,如iTOL和FigTree。它们不仅可以帮助我们调整树的布局,还能添加各种注释信息,让整棵树看起来更加生动有趣。
总的来说,系统发育树的构建与优化是一个充满挑战但也极具乐趣的过程。从序列选择到进化树的最终呈现,每一步都需要严谨的态度和细致的操作。希望我的分享能为大家提供一些参考和启发。



发表评论 取消回复